Digitale Helfer zur Beobachtung von Molekül-Gymnastik Wie man mit Software verborgene Schätze in der Chemie findet
Häm-Proteine gibt es in vielen Varianten. Sie sind wahre Alleskönner und versorgen in nahezu jedem Bereich der belebten Natur außerordentlich viele unterschiedliche Funktionen, insbesondere im biologischen Energiehaushalt. Das Zentrum in all diesen unterschiedlichen Eiweißen ist immer dasselbe Molekül. Wie aber kann eine einzelne Verbindung derartig viele Funktionen ausüben? Jens Krumsieck und Professor Martin Bröring vom Institut für Anorganische und Analytische Chemie (IAAC) der Technischen Universität Braunschweig haben ein Verfahren entwickelt, mit dem sich Antworten auf diese Frage finden lassen. Die Ergebnisse sind gerade als Very Important Paper in „Chemistry – A European Journal“ veröffentlicht worden.
Das bekannteste Beispiel für Häm-Proteine ist das Hämoglobin, der Sauerstofftransporter aus dem Blut von Säugetieren. Neben dem Hämoglobin gibt es auch viele weitere bekannte Beispiele wie die Cytochrom c Oxidase, die das Kraftwerk in der Mitochondrienmembran darstellt und den Organismus mit biologischem Strom und Wärme versorgt. Auch das Cytochrom P450 könnte Vielen bekannt sein. Mit ihm werden in unserer Leber Xenobiotika abgebaut und der Körper entgiftet.
Zentrales Molekül: Ein Eisenkomplex des Protoporphyrin IX
Das aktive Zentrum in all diesen unterschiedlichen Eiweißen ist hingegen immer dasselbe Molekül: ein Eisenkomplex des Protoporphyrin IX, die sogenannte Häm-Gruppe. Häm ist im Wesentlichen ein nahezu quadratisches und flaches 2D-Molekül. Chemisch gehört es zu den Metalloporphyrinen. Es gibt sowohl in der Natur als auch im Labor viele Variationen davon, zum Beispiel die Magnesium-haltigen Chlorophylle in grünen Pflanzen oder das Kobalt-Chlorin vom Vitamin B12.
Seit langem stellt man sich die Frage, wie eine einzelne Verbindung in der Lage sein kann, derartig viele unterschiedliche Funktionen mit einer derartig hohen Selektivität und Spezifität auszuüben. Jens Krumsieck und Martin Bröring haben dafür ein digitales Tool entwickelt: „PorphyStruct“ entdeckt besondere Muster in den Strukturen von Porphyrinoiden.
„Molekulare Gymnastik“ erkennen und quantifizieren
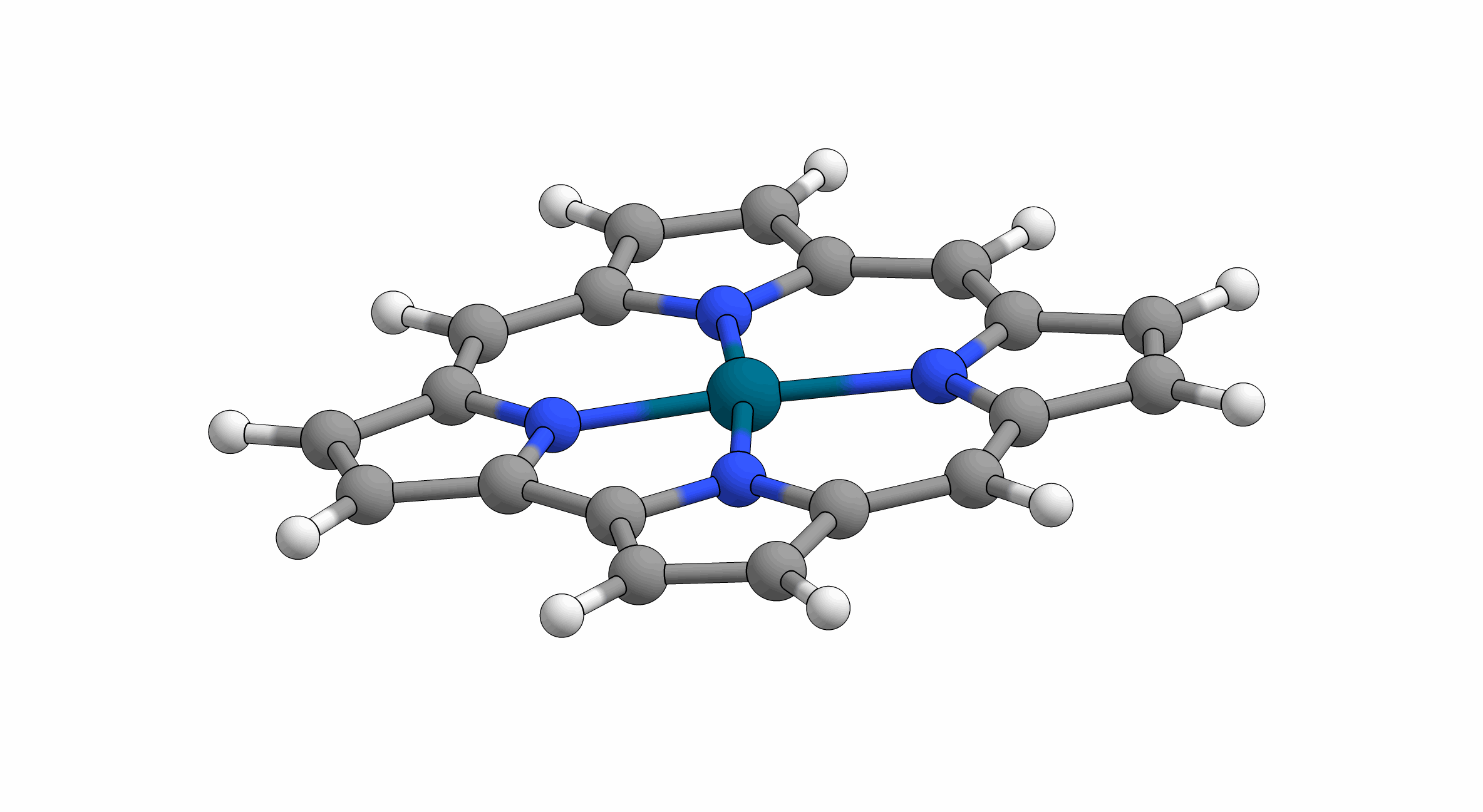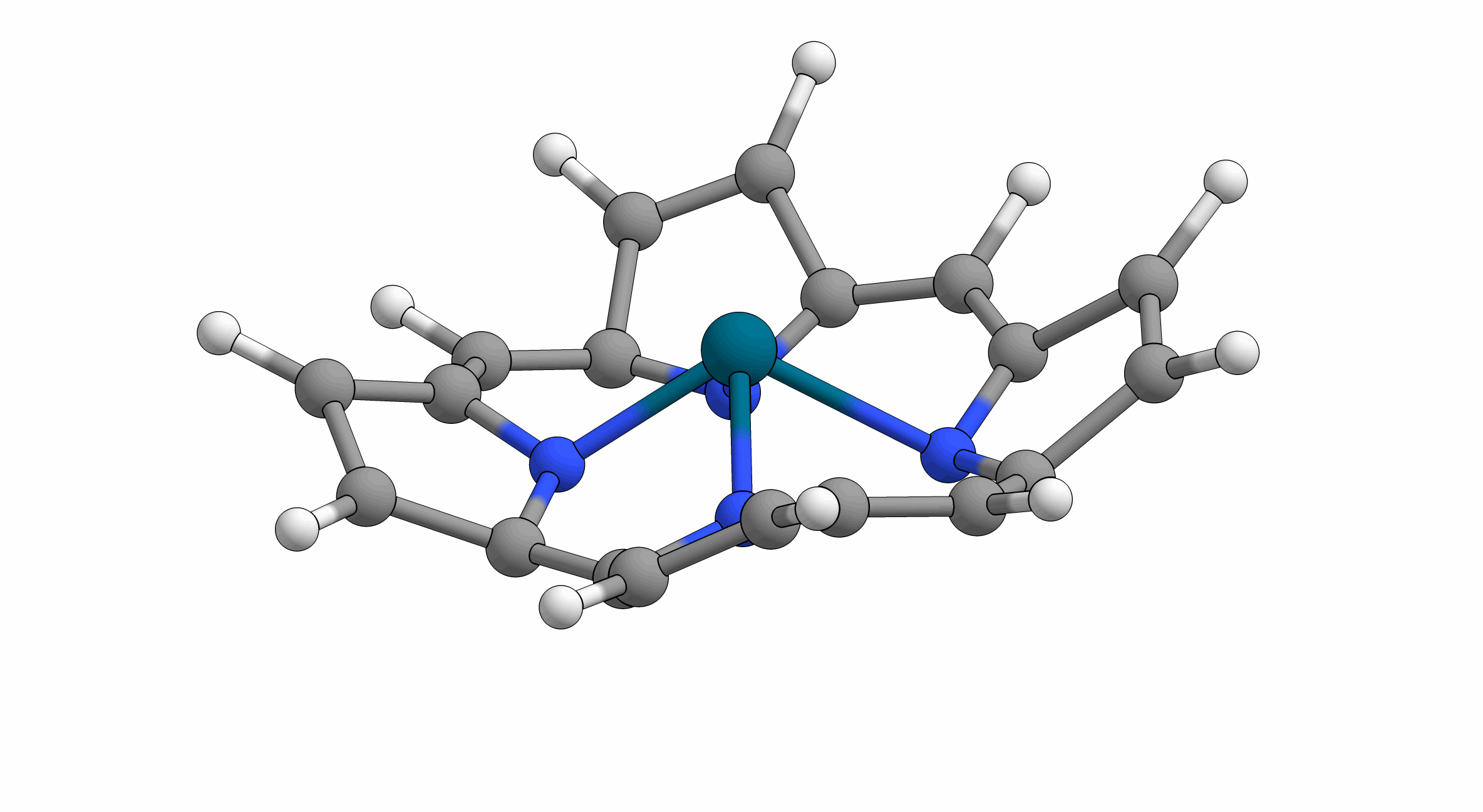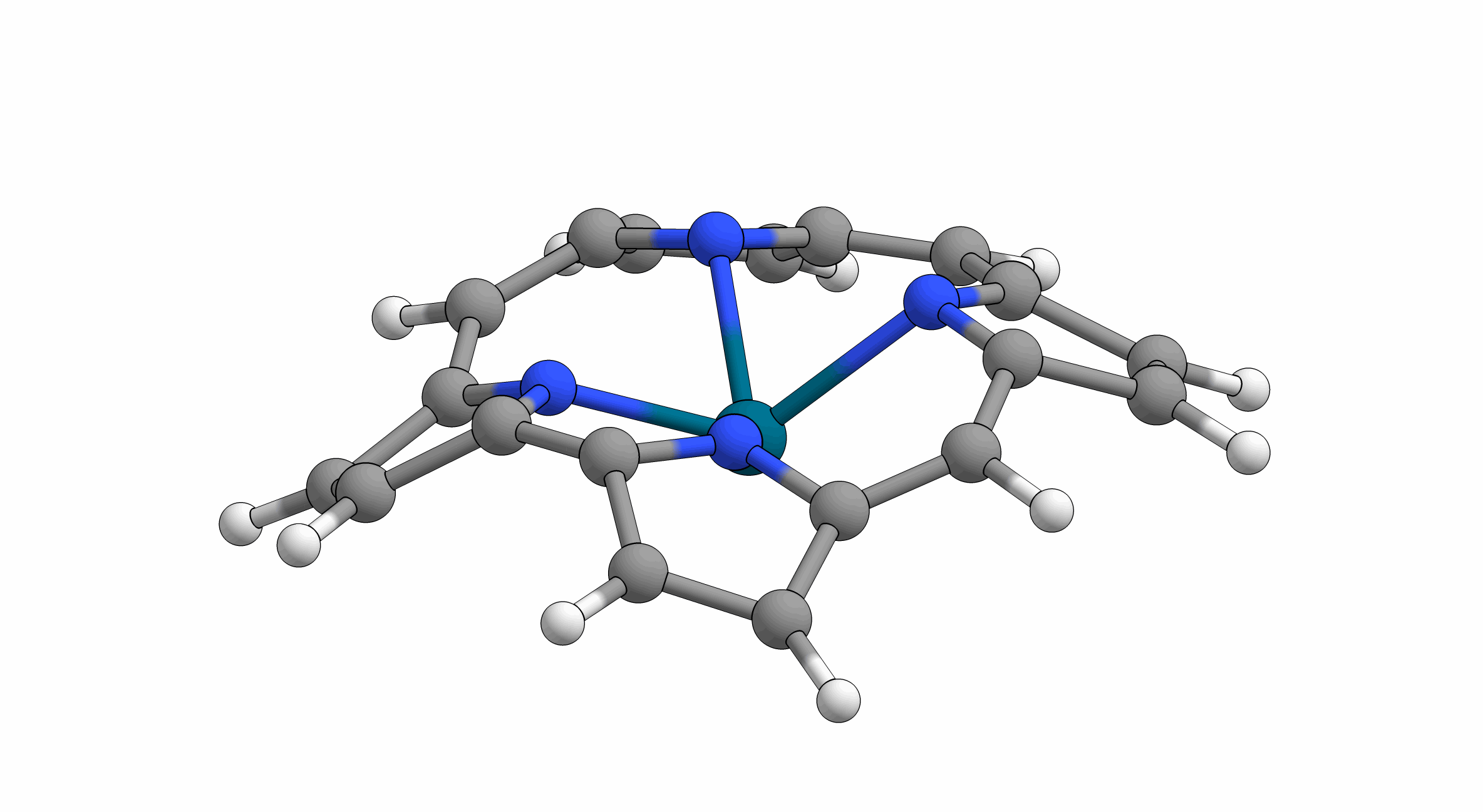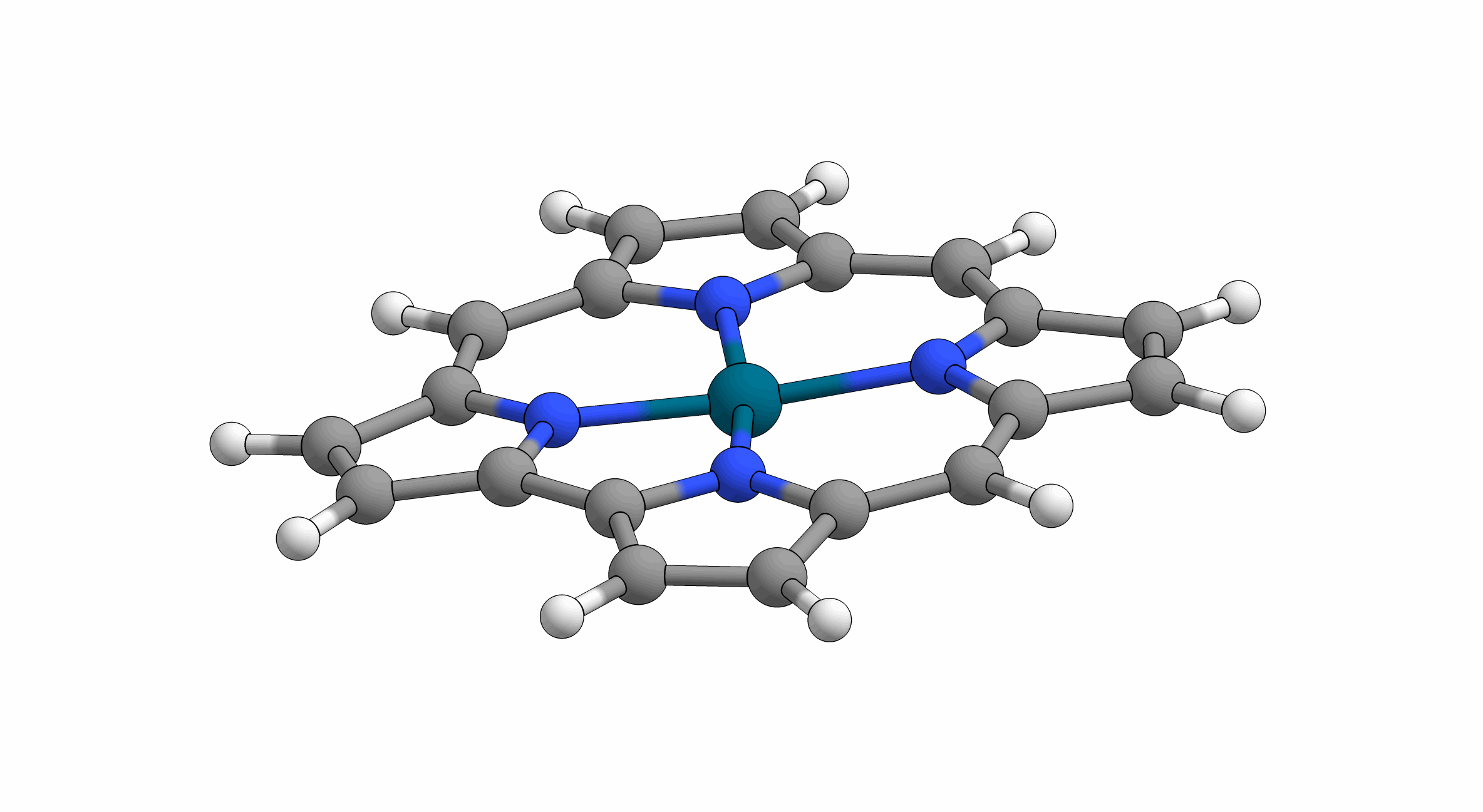
Animation: Darstellung einer „gymnastischen“ Konformationsmode (doming2). Bildnachweis: Krumsieck/Bröring/TU Braunschweig
Diese besonderen Muster sind sogenannte Konformationsmoden. Das Einnehmen dieser Moden durch die Porphyrinoide wurde jüngst von Professor Mathias Senge vom Trinity College Dublin (Irland) als „Molekulare Gymnastik“ betitelt. Porphyrinoide sind tatsächlich wahre Sportler und alles andere als unbeweglich. Sie können sich auf verschiedenste Weise dehnen und verbiegen – verbunden mit den positiven Effekten ähnlich unserer Gymnastik. Mit dieser Verbiegung verändern sich elektrische und optische Eigenschaften der Moleküle zum Teil drastisch. Dies bietet einen Erklärungsansatz dafür, wieso sich auch die Reaktivität der Häm-Gruppe in verschiedenen Proteintaschen so stark ändert.
„PorphyStruct“ ist in der Lage, diese Dehnübungen automatisch aufzuspüren, zu kategorisieren und zu quantifizieren, und zwar für Strukturen aus den unterschiedlichsten Bereichen der Wissenschaft. Damit bietet das Programm eine einfache und für Chemiker, Biologen, Ingenieure und Materialwissenschaftler leicht einsetzbare Möglichkeit, Porphyrinoide in unterschiedlichen Umgebungen quantitativ zu bewerten.
„Das Projekt ist ganz klar in der Grundlagenforschung angesiedelt“, sagt Bröring. „Aber natürlich kann ein digitales Tool auch immer sehr schnell eingesetzt werden, wenn es um die Aufdeckung von verborgenem Wissen mit Anwendungsbezug geht. Aus diesem Grunde habe wir uns auch bewusst dazu entschlossen, den Quellcode völlig frei als Open Source zugänglich zu machen.“
Ergebnis als „Very Important Paper“ eingestuft
Die Einsicht, dass die „gymnastischen“ Fertigkeiten der Porphyrinoide eine wichtige Komponente im Verständnis ihrer Reaktivität darstellen, ist nicht neu. Eine einfache Quantifizierungsmöglichkeit, die sich über die meisten bekannten Systeme erstreckt und vergleichbare Analysen ergibt, gab es hingegen bislang nicht. Der Bedarf an einer solchen Methode war groß. Meist wurden einzelne Fälle bisher in äußerst langwierigen Verfahren händisch auf ihre Verbiegungsmuster untersucht. Einer der Referees bringt dies auf den Punkt und schreibt dazu in seinem Gutachten: „This is certainly a long-awaited tool in the porphyrin community.“
Professor Bröring ergänzt: „Eigentlich war die Methode und damit auch so ein Paper für uns präparative Chemiker weit außerhalb unserer Wohlfühlzone. Aber das Risiko, mal aus einem völlig neuen Blickwinkel draufzuschauen, hat sich gelohnt. Es wird sehr viel internationale Aufmerksamkeit im Fach geben, wenn die Arbeit einmal draußen ist.“ Die Bedeutung und erwartete Resonanz wird auch die durch Klassifizierung des Braunschweiger Beitrages als „Very Important Paper“ in „Chemistry“ unterstrichen. In Journalen werden besonders herausragende wissenschaftliche Beiträge VIP klassifiziert.
„Und die Forschung ist uns auch nicht leicht gefallen“, bemerkt Krumsieck. „Dem Computer beizubringen, bestimmte Muster im dreidimensionalen Raum zu erkennen, hat sich als nicht trivial herausgestellt. Unser Ziel musste aber sein, dass die Bedienung möglichst einfach und intuitiv ist.“ Dafür müssen bestimmte Strukturmotive automatisiert erkannt werden – vor allem in der Masse von über 150 Test-Strukturen war das zusätzlich eine sehr zeitintensive Aufgabe.
Visuelles Gedankenspiel auf dem Titelblatt der Chemistry-Ausgabe

Titel der Chemistry-Ausgabe. Bildnachweis: Wiley-VCH.
Auf dem Titelblatt von „Chemistry – A European Journal“ ist die Darstellung eines Gedankenexperiments abgebildet: Was wäre, wenn Moleküle lebensgroße Entitäten wären, die man direkt mit dem bloßen Auge in futuristischen Screening-Laboren untersuchen könnte? Man könnte wie ein Fitnesstrainer überprüfen, wie gut ein Molekül die angeordneten Dehnübungen durchführt und so den Fitness-Zustand bewerten. In der Realität geht das natürlich nicht. „Da müssen wir auf die verfügbaren Werkzeuge zur Untersuchung submikroskopischer Quanten-Strukturen wie der Moleküle zurückgreifen“, sagt Professor Bröring. Und zu diesen Werkzeugen gehört nun auch „PorphyStruct“.
